Módulo 3 T2020
2 participantes
Página 1 de 1
 Módulo 3 T2020
Módulo 3 T2020
Oi, gente, boa tarde. Fiquei com algumas dúvidas sobre a atividade 3, consegui no geral fazer a atividade, mas o meu barplot ficou com os nomes de sim e não ao invés de sanhaços e melastomataceae, vi e revi os passos, mas não consegui identificar o erro, alguém poderia em ajudar? Abaixo segue o script, resultado no console do R e o gráfico que consegui fazer, desde obrigado a quem puder ajudar:
#TAREFA 1 - UTILIZANDO O ARQUIVO SANHAÇOS
#Importando dados
setwd("C:/Pasta R/Curso R/Módulo 3/Scripts e dados M3/atividade")
dir()
#Montando o objeto
sanhacos<-read.table("sanhacos.txt", header=T)
#Conferindo os dados
summary(sanhacos)
#Criando a tabela de contingência com a função table() e verificar qual variável fica mlhor em qual posição
tabelasanhacos<-table(sanhacos$Sanhaco, sanhacos$Melastomatacea)
#Olhando a tabela
tabelasanhacos
#Criando a tabela em outra ordem, só pra comparar:
tabelasanhacos2<-table(sanhacos$Melastomatacea, sanhacos$Sanhaco)
tabelasanhacos2
#Se quisermos, podemos calcular uma tabela de frequências relativas:
tabela.prop<-prop.table(tabelasanhacos, margin=1)
tabela.prop
tabela.prop2<-prop.table(tabelasanhacos, margin=1)
tabela.prop2
#margin=1 é utilizado os dados da linha para verificar a porcentagem, se eu quisesse usar a porcentagem da coluna é só colocar margin=2, é o que fiz na minha!
#fAZENDO O gráfico versão básica:
#1 TABELA SIMPLES COM GRÁFICOS EMPILHADOS
barplot(tabelasanhacos)
#2 TABELA COM GRÁFICOS SEPARADOS POR CONTA DA FUNÇÃO BESIDE, O r L~E EM ORDEM ALFABÉTICA, ENTÃO MELAST VEM PRIMEIRO Q SANHACO
barplot(tabelasanhacos, beside=T)
barplot(tabelasanhacos2, beside=T)
#Melhorando o gráfico
barplot(tabelasanhacos, beside=T, ylim=c(0, 80), las=1, xlab="Melatomatacea", ylab="Sanhaços")
CONSOLE R
> sanhacos<-read.table("sanhacos.txt", header=T)
> summary(sanhacos)
UA Sanhaco Melastomatacea
Min. : 1.0 Nao:74 Nao:52
1st Qu.:25.5 Sim:25 Sim:47
Median :50.0
Mean :50.0
3rd Qu.:74.5
Max. :99.0
> tabelasanhacos<-table(sanhacos$Sanhaco, sanhacos$Melastomatacea)
> tabelasanhacos
Nao Sim
Nao 40 34
Sim 12 13
> tabelasanhacos2<-table(sanhacos$Melastomatacea, sanhacos$Sanhaco)
> tabelasanhacos2
Nao Sim
Nao 40 12
Sim 34 13
> #Se quisermos, podemos calcular uma tabela de frequências relativas:
> tabela.prop2<-prop.table(tabelasanhacos, margin=1)
> tabela.prop2
Nao Sim
Nao 0.5405405 0.4594595
Sim 0.4800000 0.5200000
> barplot(tabelasanhacos)
> barplot(tabelasanhacos, beside=T)
> tabelasanhacos2<-table(sanhacos$Melastomatacea, sanhacos$Sanhaco)
> tabelasanhacos2
Nao Sim
Nao 40 12
Sim 34 13
> #Se quisermos, podemos calcular uma tabela de frequências relativas:
> tabela.prop<-prop.table(tabelasanhacos, margin=1)
> tabela.prop
Nao Sim
Nao 0.5405405 0.4594595
Sim 0.4800000 0.5200000
> tabela.prop2<-prop.table(tabelasanhacos, margin=1)
> tabela.prop2
Nao Sim
Nao 0.5405405 0.4594595
Sim 0.4800000 0.5200000
> barplot(tabelasanhacos)
> barplot(tabelasanhacos, beside=T)
> barplot(tabelasanhacos2, beside=T)
> barplot(tabelasanhacos, beside=T)
> legend("topleft", legend=levels(tabelasanhacos$Melatomatacea), col=cores, pch=15, title="Relação Sanhaços e Melasomataceae")
Error in tabelasanhacos$Melatomatacea :
$ operator is invalid for atomic vectors
> barplot(tabelasanhacos, beside=T, ylim=c(0, 80), las=1, xlab="Melatomatacea", ylab="Sanhaços")
>
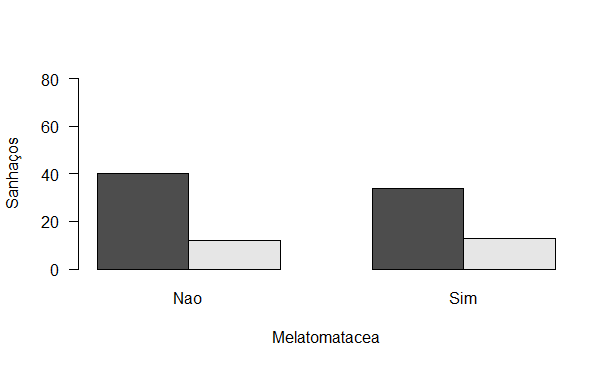
#TAREFA 1 - UTILIZANDO O ARQUIVO SANHAÇOS
#Importando dados
setwd("C:/Pasta R/Curso R/Módulo 3/Scripts e dados M3/atividade")
dir()
#Montando o objeto
sanhacos<-read.table("sanhacos.txt", header=T)
#Conferindo os dados
summary(sanhacos)
#Criando a tabela de contingência com a função table() e verificar qual variável fica mlhor em qual posição
tabelasanhacos<-table(sanhacos$Sanhaco, sanhacos$Melastomatacea)
#Olhando a tabela
tabelasanhacos
#Criando a tabela em outra ordem, só pra comparar:
tabelasanhacos2<-table(sanhacos$Melastomatacea, sanhacos$Sanhaco)
tabelasanhacos2
#Se quisermos, podemos calcular uma tabela de frequências relativas:
tabela.prop<-prop.table(tabelasanhacos, margin=1)
tabela.prop
tabela.prop2<-prop.table(tabelasanhacos, margin=1)
tabela.prop2
#margin=1 é utilizado os dados da linha para verificar a porcentagem, se eu quisesse usar a porcentagem da coluna é só colocar margin=2, é o que fiz na minha!
#fAZENDO O gráfico versão básica:
#1 TABELA SIMPLES COM GRÁFICOS EMPILHADOS
barplot(tabelasanhacos)
#2 TABELA COM GRÁFICOS SEPARADOS POR CONTA DA FUNÇÃO BESIDE, O r L~E EM ORDEM ALFABÉTICA, ENTÃO MELAST VEM PRIMEIRO Q SANHACO
barplot(tabelasanhacos, beside=T)
barplot(tabelasanhacos2, beside=T)
#Melhorando o gráfico
barplot(tabelasanhacos, beside=T, ylim=c(0, 80), las=1, xlab="Melatomatacea", ylab="Sanhaços")
CONSOLE R
> sanhacos<-read.table("sanhacos.txt", header=T)
> summary(sanhacos)
UA Sanhaco Melastomatacea
Min. : 1.0 Nao:74 Nao:52
1st Qu.:25.5 Sim:25 Sim:47
Median :50.0
Mean :50.0
3rd Qu.:74.5
Max. :99.0
> tabelasanhacos<-table(sanhacos$Sanhaco, sanhacos$Melastomatacea)
> tabelasanhacos
Nao Sim
Nao 40 34
Sim 12 13
> tabelasanhacos2<-table(sanhacos$Melastomatacea, sanhacos$Sanhaco)
> tabelasanhacos2
Nao Sim
Nao 40 12
Sim 34 13
> #Se quisermos, podemos calcular uma tabela de frequências relativas:
> tabela.prop2<-prop.table(tabelasanhacos, margin=1)
> tabela.prop2
Nao Sim
Nao 0.5405405 0.4594595
Sim 0.4800000 0.5200000
> barplot(tabelasanhacos)
> barplot(tabelasanhacos, beside=T)
> tabelasanhacos2<-table(sanhacos$Melastomatacea, sanhacos$Sanhaco)
> tabelasanhacos2
Nao Sim
Nao 40 12
Sim 34 13
> #Se quisermos, podemos calcular uma tabela de frequências relativas:
> tabela.prop<-prop.table(tabelasanhacos, margin=1)
> tabela.prop
Nao Sim
Nao 0.5405405 0.4594595
Sim 0.4800000 0.5200000
> tabela.prop2<-prop.table(tabelasanhacos, margin=1)
> tabela.prop2
Nao Sim
Nao 0.5405405 0.4594595
Sim 0.4800000 0.5200000
> barplot(tabelasanhacos)
> barplot(tabelasanhacos, beside=T)
> barplot(tabelasanhacos2, beside=T)
> barplot(tabelasanhacos, beside=T)
> legend("topleft", legend=levels(tabelasanhacos$Melatomatacea), col=cores, pch=15, title="Relação Sanhaços e Melasomataceae")
Error in tabelasanhacos$Melatomatacea :
$ operator is invalid for atomic vectors
> barplot(tabelasanhacos, beside=T, ylim=c(0, 80), las=1, xlab="Melatomatacea", ylab="Sanhaços")
>
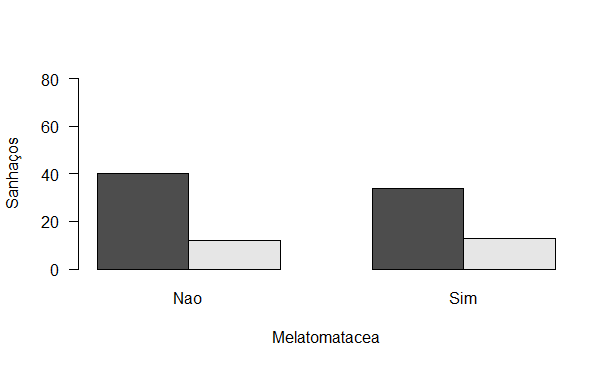
nani2410- Mensagens : 8
Data de inscrição : 31/03/2020
 Re: Módulo 3 T2020
Re: Módulo 3 T2020
Então, há um pouquinho de confusão em relação aos eixos. O eixo y é o número de observações, independente da escolha da versão 1 ou dois de tabelasanhacos, ok?
O eixo x será uma das variáveis (ou sanhalgos ou melastomatáceas), e os valores dos eixos estão corretos como sim ou não, só é importante sabermos para qual das duas variáveis estamos olhando.
Também há um errinho na tentativa de se criar a legenda, pois você usa o objeto tabelasanhacos para os níveis da variável, e neste caso deveria usar o objeto com os dados originais.
Refazendo seu script, veja como fica:
No fim, este caso é um tanto confuso mesmo, pois as duas variáveis usadas são codificadas como sim ou não, então fica bem fácil se perder. A dica que eu dou é usar o summary() dos dados e comparar os totais com o que você vê no objeto tabelasanhacos, pois assim fica mais fácil saber o que está se interpretando e decidir pelo gráfico que achar melhor.
O eixo x será uma das variáveis (ou sanhalgos ou melastomatáceas), e os valores dos eixos estão corretos como sim ou não, só é importante sabermos para qual das duas variáveis estamos olhando.
Também há um errinho na tentativa de se criar a legenda, pois você usa o objeto tabelasanhacos para os níveis da variável, e neste caso deveria usar o objeto com os dados originais.
Refazendo seu script, veja como fica:
- Código:
sanhacos<-read.table("sanhacos.txt", header=T, stringsAsFactors = T)
summary(sanhacos)
tabelasanhacos<-table(sanhacos$Sanhaco, sanhacos$Melastomatacea)
tabelasanhacos
tabelasanhacos2<-table(sanhacos$Melastomatacea, sanhacos$Sanhaco)
tabelasanhacos2
cores=c("red4", "green4")
barplot(tabelasanhacos, beside=T, xlab="Melastomataceas", ylab="Observacoes", ylim=c(0, 50), col=cores)
legend("topright", legend=levels(sanhacos$Sanhaco), pch=15, title="Sanhacos", col=cores)
barplot(tabelasanhacos2, beside=T, xlab="Sanhacos", ylab="Observacoes", ylim=c(0, 50), col=cores)
legend("topright", legend=levels(sanhacos$Sanhaco), pch=15, title="Melastomatacea", col=cores)
No fim, este caso é um tanto confuso mesmo, pois as duas variáveis usadas são codificadas como sim ou não, então fica bem fácil se perder. A dica que eu dou é usar o summary() dos dados e comparar os totais com o que você vê no objeto tabelasanhacos, pois assim fica mais fácil saber o que está se interpretando e decidir pelo gráfico que achar melhor.
 Tópicos semelhantes
Tópicos semelhantes» Atividade do módulo 1
» Módulo Extra 6
» Atividade Modulo 3- Duvidas e comentários
» Atividade Módulo-3
» Dúvidas Módulo 2 - Turma 2020
» Módulo Extra 6
» Atividade Modulo 3- Duvidas e comentários
» Atividade Módulo-3
» Dúvidas Módulo 2 - Turma 2020
Página 1 de 1
Permissões neste sub-fórum
Não podes responder a tópicos|
|
|
